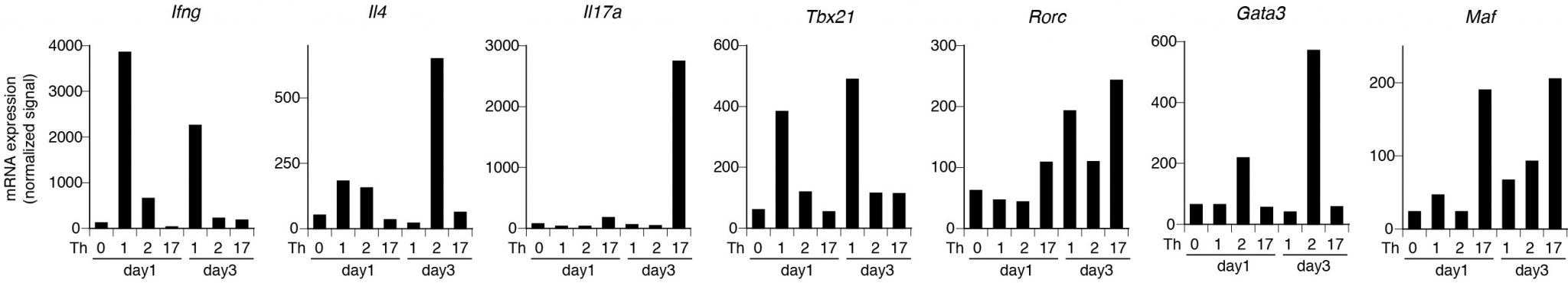
さて、話が大分脱線したので戻りますと、Th1, Th2, Th17細胞のトランスクリプトーム解析の話でした。結果を見ると、それぞれのサブセットで、代表的なサイトカインであるIFN-γ、IL-4, IL-17が増えていますしTh1細胞では確かにマスターレギュレーター転写因子(くどういようですが今はあまりそう呼ばないかもしれません。)のT-bet (Tbx21)の発現が増えています。Th2細胞ではGata-3の発現が増えていますし、Th17細胞でも過去の報告にあったRORγtをコードするRorcや、その仲間であるRoraの発現が増えています。実験自体は上手くいっていそうです。それではRorcやRoraの他に、Th17細胞で誘導される転写因子はあるでしょうか?・・・ありました。Maf遺伝子です。これがコードする転写因子はc-Mafです。あれ?c-Maf ?? これはTh2細胞分化に重要な転写因子のはずだが・・・。
Th2細胞よりもTh17細胞の方が発現が高いように見えます。これは予想していなかった結果です。しかしデータ自体ははっきりしていましたので、c-Mafのノックアウトマウスとトランスジェニックマウスを筑波大の高橋智先生に御供与いただいて解析することにしました。
ここでまた話が逸れていきますが、今回Th1, Th2, Th17細胞のそれぞれ分化1日目と3日目、そしてニュートラルな条件で培養した(いわばTh0)細胞の1日目、の合わせて7サンプルをトランスクリプトーム解析した訳です。それをどのように表現するのかというところが悩みどころでした。遺伝子の一つ一つに注目すれば、ただのヒストグラムでも十分に分かりやすく表現できると思います(図参照)。
しかし、遺伝子はそれぞれが相互作用しているものですし、GeneChipには数万の(重複あり)遺伝子が載っています。7つのサンプルなので、次元で表すと数十万次元ということになります。それをどのように表現できるでしょうか。分化条件が単一(たとえばTh1分化のみ)であれば時系列でもヒートマップという方法で表現できます。7サンプルであってもヒートマップを使えなくもないのですが、私にとっては必ずしも分かりやすい表現方法ではありませんでした。色々悩んだ末にたどり着いた方法は「因子分析」という方法です。
佐藤 浩二郎
私的免疫学ことはじめ (39)← Prev Next →私的免疫学ことはじめ (41)